我如期来更新啦!!!聚类算法是很常用的一种算法,不过最常见的就是KMeans了,虽然很多人都会用,不过讲道理,如果是调用现成机器学习库里面的KMeans的话,我敢保证90%的人答不上来具体的是什么算法。相信我,库里的KMeans跟教科书讲的那个随机取初始点的KMeans不是一个算法哟~
因为KMeans依赖K,但是我怎么知道K要用多少呢?另外,KMeans受限于算法本身,对于球状的数据效果较好,但是不规则形状的就不行了。这种情况下,相对而言,基于密度的聚类算法就比较好用了。sklearn里面现在是放了一个DBSCAN,下一版会更新OPTICS。刚好最近都用了,这里把DBSCAN跟OPTICS算法复现一遍。
DBSCAN
DBSCAN算法的原始论文是96年的这篇《A Density-Based Algorithm for Discovering Clusters in Large Spatial Database with Noise》。
DBSCAN是一种基于密度的聚类算法,也就是说,密度高的区域自动聚成一类。这样一来,我们就避免了人为去设定群组数量的问题,算法可以自动发现群组数量。另外用这种方法,如果一个sample不在高密度区域,就有可能被判定为异常值,那么也可以拿来作为异常值检验的方法。
DBSCAN的思路非常简单,有两个参数,一个是\(\varepsilon\),另一个是minimum points。这里首先定义DBSCAN的几个核心概念,一个是\(\varepsilon\)-neighborhood,另一个是core object,还有就是reachable。
首先是\(\varepsilon\)-neighborhood。DBSCAN开始的时候会随机选取一个初始点,然后按照\(\varepsilon\)的距离寻找临近点,这些点的集合叫做\(\varepsilon\)-neighborhood。参数里面的minimum points就是限定这个点的集合的。minimum points限定了这个集合最小需要包含多少样本点,\(\varepsilon\)则是限定了要用多大的范围去框定这些样本点。这里有个小细节要注意,那就是,算neighborhood的时候,中心点自己是算进来的。
那么这样圈完一波neighborhood后,我们会将符合样本数量大于等于minpts的中心点叫做core object。而core object跟这些neighbor就是reachable的。
然后为了让算法运作起来,只要neighborhood这个集合里面有点,我们就不断重复这样圈地的动作,然后把中心点从集合中拿掉,直到neighborhood为空。
那我们很自然就会想到,一定会圈到一些点,它们不是core object,但是也在集合里面。这些点我们叫做border,也就是说,这些点是这个类的边界了。那么我们就很自然会想到,也有一些点压根不在neighborhood里面,也不是core的点,这些点就是noise。那既然样本点多了两个状态,reachable的情况也就变得多了,如果是直接可以在neighborhood里面找到的,我们叫做directly-reachable;如果通过neighborhood一层层找,最后找到的,我们叫density-reachable。
可以看图说话:

这个图里面,A是core,B、C是border,N是noise。A跟B、C都是density-reachable。但是有没有发现,B是没法返回去找到A的。所以这种reachable是有方向的。
如果对SNA有点了解的朋友就知道,就是一度人脉和N度人脉,但是是一个有向图。
那么算法思路理清了,代码就好写了,这里我就用最常用的欧氏距离了。有人可能会想,如果还是欧氏距离,那跟KMeans还有什么分别,都是画圈圈嘛!请回想一个微积分,只要圈画的够小,就能做出各种形状来。下面是核心部分的代码,详细的可以去看我的notebook。
1 | def _euclidean_dist(p, q): |
OPTICS
那么DBSCAN本身是一个非常牛逼的算法,它解决了我们找K的问题,这样在海量群组的时候,我们不用像KMeans一样去到处尝试K的大小。但是DBSCAN有个问题,那就是这个算法只能检测一个密度。换句话说,如果现在存在一个数据集有两个类,一个类是方差小的,一个类是方差大的。且这两个群组离得不算太远。如果我们为了照顾方差大的群组将eps设得很大,minpts设得很小,那么可能把两个类聚在一起。反过来,我们就可能找不到方差大的类。
那么问题来了,有没有办法量化这个距离呢?三年后,同一组作者在DBSCAN的基础上进化出了OPTICS算法。
既然是DBSCAN的进化版,所以很多概念上都是互通的,只是OPTICS算法多了几个概念,一个是core-distance,一个是reachability-distance。
我们知道,DBSCAN是不断跑马圈地的一个过程,但是我们很直观想就知道,有些密度大的地方,可能不需要\(\varepsilon\)那么大的范围就可以圈到minpts个样本,所以在OPTICS算法里面,我们将满足minpts这么多样本点的\(\varepsilon'\)叫做core-distance。而reachability-distance就是中心点与临近点的距离,但是,如果临近点落在\(\varepsilon'\)内,reachability-distance就用core-distance来替代。如下图:

那么用OPTICS的时候我们就需要定义两个列表,一个是seeds,一个是ordered result。seeds就是我们每一轮迭代时候的候选列表,而ordered result就是最终的结果。
具体的过程是这样的。我们先找到一个点,然后一样跑马圈地,接着计算reachability-distance,然后放到seeds里面从小到大排序。每次取第一个seed出来继续圈地,把被取出来的点以及这个点的reachability distance存在ordered result里面。接着就跟DBSCAN一样,不断重复,直到neighborhood为空。这样做的好处就是,我们可以量化评估每个群的密度大小。效果如下图:

那么我们又会想到,有些seeds里面的点可能随着核心点的移动,reachability distance会不断变小。因为A的core distance里可能是B,而C不在A的core distance里面,但是C在B的core distance里面。如果第一个处理的点是A,第二个处理的点是B,那C其实还是很核心的一个点。那这种时候我们就要跟一它开始的reachability distance做比较,如果新的reachability distance比原来的小,就把原来的值替换掉。
那么废话不多说,上代码,很多地方跟DBSCAN是可以复用的,我就放了一些OPTICS的核心部分,老规矩,详细见notebook:
1 | def _eps_neighborhood(data, point_id, eps): |
OPTICS的优点就是,不管是什么形状的密度,基本上都可以把这个凹槽给跑出来,但是问题就是最后的这个抽取群组小算法。目前我还没找到一个比较好的方法来自动抽取,如果是按照论文里面的分层抽取,我试过会抽的太细,如果是按照论文里面的DBSCAN来抽,就是我实现的这个,不过是一刀切的方式,太复杂的样本效果就不好了。目前还在探索用其他平滑方法来替代,有突破再来更新。
试了两种平滑方式,最后的做法是,先用一维高斯平滑,然后用max filter抹掉因为高斯平滑最大值向左漂的问题。
HDBSCAN
然后又发现了一个新的聚类算法,类似OPTICS算法,叫HDBSCAN,顾名思义,就是H就是hierarchical。事实上就是OPTICS算法的一个改进版本。
与OPTICS类似,HDBSCAN一样要算core distance和reachability distance。这个算法跟OPTICS是一样的。但是reachability distance的公式是:\(d_{\text{reach}-k}(a, b) = \max\{\text{core}_k(a), \text{core}_k(b), d(a, b)\}\),这里的\(d(a, b)\)是两个点用距离公式算的距离。如下图所示:
蓝绿两点直接的reachability distance就是绿点的core distance,红绿两点的就是红绿两点的距离。
那么这样一来,就可以得到每个点之间的reachability distance。然后就可以用这个距离作为权重来绘制一个带权重的连接图。Beyond Hartigan Consistency这篇论文认为,无论是那种概率密度分布,这种相互距离可以更好的表示单链接聚类的层次结构。
然后可以用Prim算法构建最小生成树,然后转化为层次结构,就类似层次聚类了。这个可以参考一下基于最小生成树的层次聚类。然后通过设定最小簇大小,对这个树进行剪枝。也就从下面的第一个图变成第二个图:
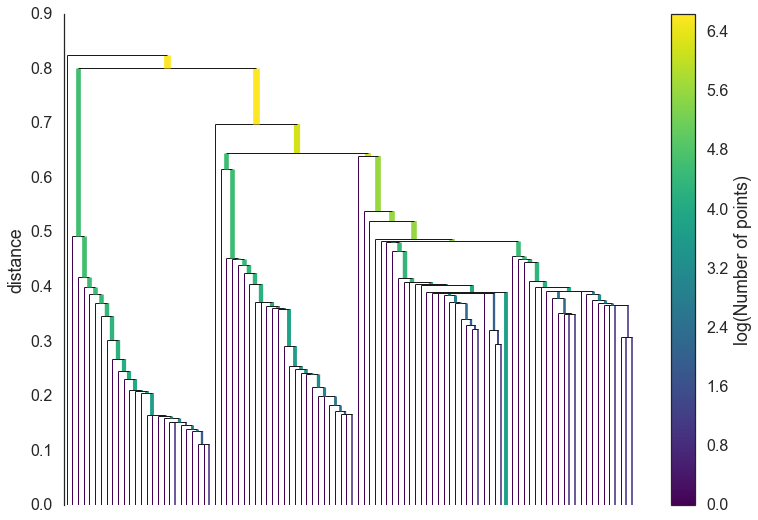

之后就是抽取簇。上面剪枝后的树其实已经有了结果,但是我们希望抽的簇可以自动抽取,且是稳定的。因此我们需要一种度量方式来衡量簇的稳定性。我们定义一个值\(f(x) = \lambda = \frac{1}{\text{distance}_{\text{core}}}\)。这里的distance就是一个点。所以用\(\lambda_{\text{birth}}\)和\(\lambda_{\text{death}}\)分别表示簇生成和簇分裂时候的\(\lambda\)大小,也就是\(\lambda_{\text{min}}C_i\)和\(\lambda_{\text{max}}C_i\),那么簇的稳定性就是\(S = \sum_{p \in \text{cluster}} (\lambda_p - \lambda_{\text{birth}})\),\(\lambda_p = \lambda_{\text{max}}(x, C) = \min(f(x), \lambda_{\text{max}}C)\)每轮优化的方向就是让这个稳定性最大,同时满足最小簇大小。
那么HDBSCAN跟DBSCAN比的话,DBSCAN实际上就是上面剪枝前的hierarchical tree切一刀,而HDBSCAN会自适应地去寻找合适的划分。
